Spésifikasi deteksi
Dina kalolobaan kasus, tujuan desain primer nyaéta pikeun maksimalkeun spésifisitas PCR.Ieu ditangtukeun ku pangaruh leuwih atawa kurang bisa diprediksi tina loba variabel.Hiji variabel penting nyaéta runtuyan dina 3′ tungtung primer.
Anu penting, tes PCR anu dirancang pikeun spésifisitas langkung dipikaresep pikeun ngajaga efisiensi anu luhur dina rentang dinamis anu lega, sabab assay henteu ngahasilkeun produk amplifikasi non-spésifik, ku kituna bersaing sareng réagen PCR atanapi ngahambat réaksi amplifikasi utama.
Tangtosna, dina sababaraha kasus, spésifisitas sanés anu paling penting, contona, nalika tujuanana pikeun ngitung patogén anu raket tapi béda, desain khusus, optimasi sareng standar verifikasi diperyogikeun.
Kurva lebur mangrupikeun metode standar pikeun meunteun spésifisitas amplikon, sahenteuna dina hal naha ngagedékeun hiji udagan.Sanajan kitu, eta kudu emphasized yén kurva lebur bisa nyasabkeun sabab, contona, aranjeunna bisa kapangaruhan ku épék digabungkeun tina primers suboptimal sarta konsentrasi template low.
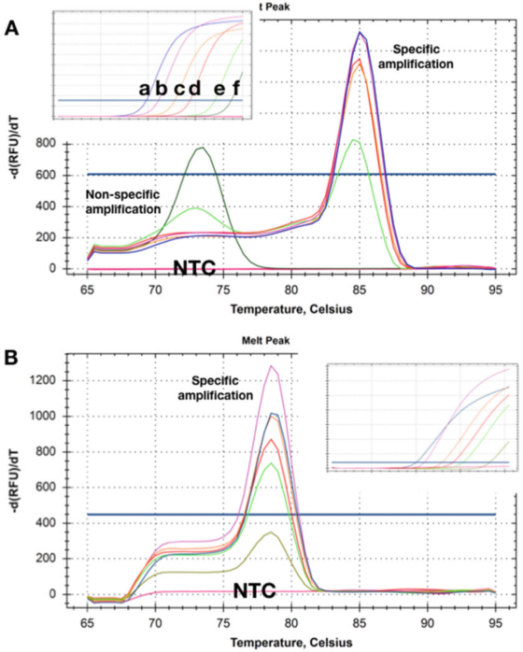
P5 |Kurva lebur nunjukkeun pergeseran Tm anu dicandak tina dua deteksi jumlah anu béda tina dua DNA target.
A. Dina konsentrasi luhur (ad)), euweuh dimer Primer atra sanggeus pangukuran qPCR réngsé.Salaku konsentrasi template nurun ka 50 salinan (e), produk non-spésifik mimiti muncul tur jadi hijina produk dina konsentrasi panghandapna (f).
B. Tés dirékam Tms sarua dina sakabéh konsentrasi target, sarta euweuh dimer Primer atra sanajan dina konsentrasi panghandapna (5 salinan).Nalika ngagunakeun dua metode deteksi ieu, teu aya produk amplifikasi anu dideteksi di NTC.
P5 nembongkeun kurva disolusi diala ku sampel nu citakan hadir dina konsentrasi béda.P 5a nunjukeun yen dina dua konséntrasi panghandapna, Tms produk amplifikasi non-spésifik dihasilkeun leuwih handap tina amplicons husus.
Jelas, metode deteksi ieu teu tiasa dipercaya pikeun ngadeteksi target anu aya dina konsentrasi rendah.
Narikna, NTCs, nyaéta, sampel tanpa DNA pisan, teu ngarekam (non-spésifik) produk amplifikasi, nunjukkeun yén latar tukang DNA génomik bisa ilubiung dina amplifikasi non-spésifik/polimérisasi.
Kadangkala primers tukang sapertos na Gedekeun non-spésifik teu bisa remedied, tapi mindeng mungkin mendesain metoda deteksi nu teu boga Gedekeun non-spésifik dina sagala konsentrasi template na NTC (P 5b).
Di dieu, sanajan ngarékam amplifikasi konsentrasi target ku Cq 35 bakal ngahasilkeun kurva disolusi husus.Nya kitu, NTCs teu némbongkeun tanda-tanda amplifikasi non-spésifik.Sakapeung, paripolah deteksi tiasa gumantung kana alkohol indung, sareng ngan ukur amplifikasi non-spésifik anu dideteksi dina komposisi panyangga anu tangtu, anu tiasa aya hubunganana sareng konsentrasi Mg2+ anu béda.
Stabilitas deteksi
Optimasi Ta mangrupikeun léngkah anu mangpaat dina verifikasi empiris sareng prosés optimasi deteksi qPCR.Eta nyadiakeun indikasi langsung tina kakuatan tina susunan Primer ku némbongkeun suhu (atawa rentang suhu) nu ngahasilkeun Cq panghandapna tanpa amplifying NTC.
Bedana dua nepi ka opat kali lipet dina sensitipitas bisa jadi teu penting pikeun jalma kalawan éksprési mRNA tinggi, tapi pikeun tés diagnostik, éta bisa hartosna béda antara hasil négatip positif jeung palsu.
Sipat Ta tina primers qPCR bisa rupa-rupa pisan.Sababaraha tés teu pisan mantap, sarta lamun maranéhna teu dipigawé dina nilai Ta optimal tina primers, aranjeunna bakal gancang ambruk.
Ieu penting sabab jenis deteksi ieu mindeng jadi masalah di dunya nyata, sarta purity sampel, konsentrasi DNA, atawa ayana DNA séjén bisa jadi teu optimal.
Salaku tambahan, jumlah salinan target tiasa rupa-rupa, sareng réagen, parabot plastik, atanapi alat tiasa bénten sareng anu dianggo nalika nyetél tés.
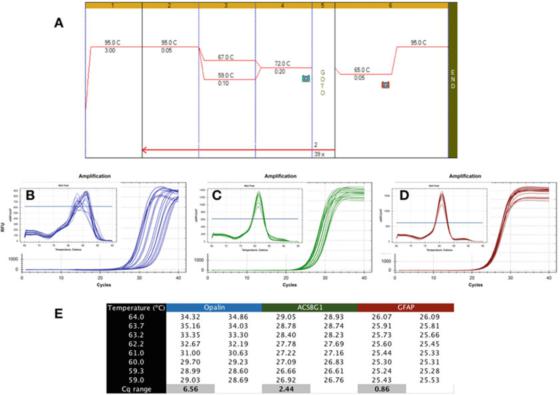
P6|Grédién hawa nunjukkeun bédana kakuatan deteksi PCR.
A. Anggo mastermix Sensifast SYBR Bioline (nomer katalog BIO-98050) pikeun ngalakukeun PCR dina cDNA anu disiapkeun tina RNA otak manusa.
B. Paké instrumen CFX qPCR Bio-Rad pikeun ngarekam peta amplifikasi jeung kurva disolusi of apalene (NM_033207, F: GCCATGGGAGGAAAGTGACAGACC, Urang Sunda: CTCATGTGTGGGTGATCTCCTAGG).
C. grafik amplifikasi jeung kurva lebur ACSBG1 (NM_015162.4, F: CTACACTTCCGGCACCACTGG, Urang Sunda: GTCCACGTGATATTGTCTTGACTCAG).
D. grafik amplifikasi jeung kurva disolusi of GFAP (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, Urang Sunda: CGAACCTCCTCCTCGTGGATCTTC).
E. Cqs dirékam dina suhu annealing béda, némbongkeun bédana dina Cq dirékam dina gradién hawa 7C.
P 6 nembongkeun hasil has tina hiji test pikaresepeun, dimana qPCR dipigawé maké Tas gradién antara 59C jeung 67C (P 6a), ngagunakeun primers pikeun tilu gén otak-spésifik manusa.
Ieu bisa ditempo tina grafik amplifikasi yén primers Opalin jauh ti idéal sabab rentang Ta optimal maranéhanana pohara heureut (Gambar 6b), nyaeta, Cqs lega dispersed, hasilna Cqs nyata dibandingkeun Cqs Low optimal maranéhanana.
Métode deteksi ieu teu stabil sareng tiasa ngakibatkeun amplifikasi suboptimal.Ku alatan éta, pasangan primers ieu kudu didesain ulang.Sajaba ti éta, analisis kurva lebur (inset) nunjukeun yen spésifisitas métode deteksi ieu ogé bisa jadi masalah, sabab kurva lebur unggal Ta béda.
Metoda deteksi ACSBG1 ditémbongkeun dina P 6c leuwih kuat batan métode deteksi Opalin luhur, tapi masih tebih ti idéal, sarta eta kamungkinan yén éta bisa ningkat.
Najan kitu, urang ngantebkeun yén euweuh sambungan diperlukeun antara ketahanan sarta spésifisitas, sabab kurva disolusi dihasilkeun ku métode deteksi ieu nembongkeun nilai puncak sarua dina sakabéh Tas (inset).
Di sisi anu sanés, uji kateguhan langkung toleran, ngahasilkeun Cq anu sami dina rupa-rupa Tas, sapertos dina uji GFAP anu dipidangkeun dina P 6d.
Beda dina Cqs diala dina rentang sarua 8 darajat Celsius kirang ti 1, sarta kurva disolusi (inset) confirms ciri deteksi dina rentang suhu ieu.Eta sia noting yén diitung Tas jeung rentang Ta sabenerna bisa jadi pohara béda.
Aya loba tungtunan dirancang pikeun mantuan peneliti ngarancang primers efisien, lolobana nu dumasar kana aturan lila-ngadegkeun jeung loba perhatian geus dibayar ka 3′tungtung primers.Hal ieu mindeng dianjurkeun pikeun ngawengku hiji G atawa C dina tungtung 3 'na dua G atawa C basa (GC clamp), tapi teu leuwih ti dua tina 5 basa panungtungan.
Dina prakték, aturan ieu bisa pituduh panalungtik, tapi maranéhna teu merta bener dina sagala kaayaan.
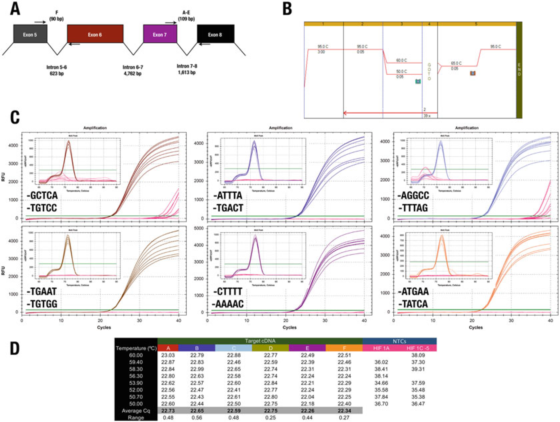
P7 |3′ tungtung primer boga pangaruh saeutik kana spésifisitas atawa efisiensi.
A. Posisi primer pikeun gén HIF-1α (NM_181054.2) manusa.
B. Paké Agilent Sarwa III SYBR Héjo indung Likur (Cat. No. 600882) pikeun ngagedékeun genep item tés.
C. grafik amplifikasi jeung kurva lebur dirékam ku instrumen CFX qPCR Bio-Rad urang jeung primers 3′tungtung.NTCs ditémbongkeun beureum.
D. Cqs catetan unggal item tés
Contona, hasil dina P 7 contradicts aturan 3'tungtung.Sadaya desain ngahasilkeun hasil anu sami, ngan ukur dua kombinasi primer anu ngarah kana amplifikasi non-spésifik di NTC.
Najan kitu, urang teu bisa ngarojong pangaruh klip GC, sabab dina hal ieu, ngagunakeun A atanapi T salaku maksimum 30 basa teu ngurangan spésifisitas.
Test C, dimana F primer ends di GGCC, teu catetan Cqs di NTCs, nunjukkeun yen salah sahiji meureun hoyong ulah runtuyan ieu dina 30-tungtung.Urang ngantebkeun yén hiji-hijina jalan pikeun nangtukeun pangalusna runtuyan 3′ahir pasangan Primer mangrupa evaluate sababaraha primers calon ékspériméntal.
Éféktivitas amplifikasi
Nu penting, sanajan deteksi PCR non-spésifik can pernah jadi spésifik, efisiensi amplifikasi bisa disaluyukeun jeung maksimal dina sababaraha cara ku cara ngarobah énzim, inuman alkohol, aditif, jeung kaayaan siklus.
Pikeun meunteun efisiensi deteksi PCR, langkung saé ngagunakeun éncér séri 10 atanapi 5 kali asam nukléat target, nyaéta, "métode kurva standar".
Upami amplikon PCR atanapi target DNA sintétik dianggo pikeun ngahasilkeun kurva standar, éncér séri target ieu kedah dicampur sareng jumlah DNA latar anu konstan (sapertos DNA génomik).
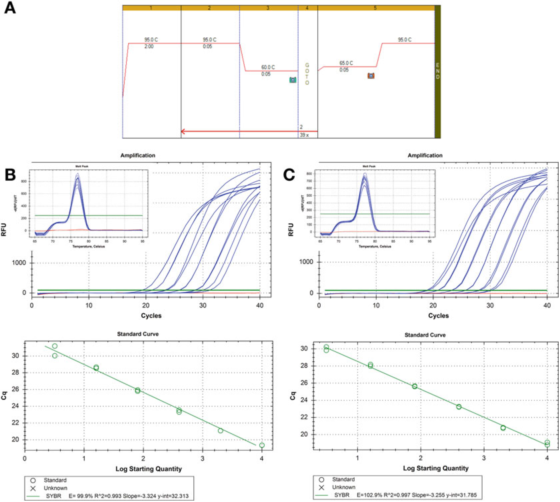
P8 |Kurva éncér pikeun ngévaluasi efisiensi PCR.
A. Paké primers pikeun HIF-1: F: AAGAACTTTTAGGCCGCTCA jeung R: TGTCCTGTGGTGACTTGTCC na Agilent urang Sarwa III SYBR Héjo mastermix (nomer katalog 600882) pikeun PCR jeung kaayaan kurva lebur.
B. 100 ng RNA ditranskripsi balik, diluted 2 kali, sarta sampel cDNA diluted serial diluted 5 kali nepi ka 1 ng DNA génomik manusa.Kurva lebur ditémbongkeun dina inset.
C. Réaksi RT, éncér, sareng éncér séri diulang pikeun sampel cDNA kadua, sareng hasilna sami.
P 8 nembongkeun dua kurva baku, ngagunakeun métode deteksi sarua dina dua sampel cDNA béda, hasilna efisiensi sarua, ngeunaan 100%, sarta nilai R2 oge sarupa, nyaeta, darajat fit antara data eksperimen jeung garis regression atawa data Gelar linearity.
Dua kurva baku anu comparable, tapi teu persis sarua.Upami tujuanna nyaéta pikeun ngitung targét sacara akurat, éta kedah diperhatoskeun yén henteu katampi masihan itungan nomer salinan tanpa ngajelaskeun kateupastian.
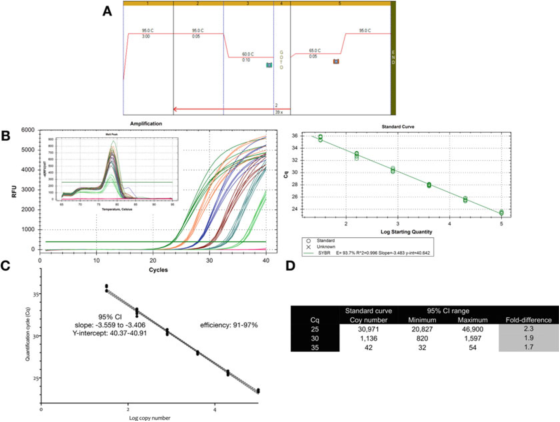
P9 |Kateupastian pangukuran pakait sareng kuantifikasi nganggo kurva standar.
A. Paké primers pikeun GAPDH (NM_002046) nedunan PCR jeung kaayaan kurva lebur.F: ACAGTTGCCATGTAGACC sareng R: TAACTGGTTGAGCACAGG sareng mastermix Sensifast SYBR Bioline (nomer katalog BIO-98050).
B. Bagan amplifikasi, kurva lebur sareng kurva standar dirékam nganggo instrumen qPCR CFX Bio-Rad.
C. grafik kurva baku sarta 95% interval kapercayaan (CI).
D. Jumlah salinan jeung interval kapercayaan 95% tina tilu nilai Cq diturunkeun tina kurva éncér.
P 9 nunjukkeun yén pikeun tés anu dioptimalkeun, variabilitas bawaan tina kurva standar tunggal sakitar 2 kali (interval kapercayaan 95%, minimum dugi ka maksimal), anu tiasa janten variabilitas pangleutikna anu tiasa dipiharep.
Produk patali:
waktos pos: Sep-30-2021